데이터 자체의 값들만으로 분류가 필요한 경우가 있다.
예를 들면, 한 사람이 아픈 증상이 있어서 병원을 찾았다. 의사는 진단해보고 이 사람이 환자라고 판단했다. 하지만 그 사람이 가진 유전체 분석을 해보니 이 사람은 정상인이었다. 이러한 경우 의사가 판단한 phenotype에 의존하지 않고 데이터만으로 분석을 진행해야 할 경우가 생긴다.
Unsupervised learning이라고 하며, t-SNE, PCA, UMAP이 있다.
추가로 point 사이의 거리에 따라 cluster해주는 k-means clustering과 Spectral clustring도 진행해 보았다.

위 그래프에서 phenotype과 data driven clustering의 결과는 상당히 다르게 보인다. 이때는 QC를 통해 feature를 선별하여 noise를 제거해야 할 것이다.
* K-means clustering : Random한 포인트들을 먼저 찍은 다음, 가까이 있는 포인트들과의 거리를 계산하여 그룹의 중심을 찾아가는 방식.
* Spectral clustering : 두 포인트를 임의로 잡아서 선을 긋고, 선으로 나누어진 그룹의 유사도(by affinity)를 계산하면서 분류하는 방식.
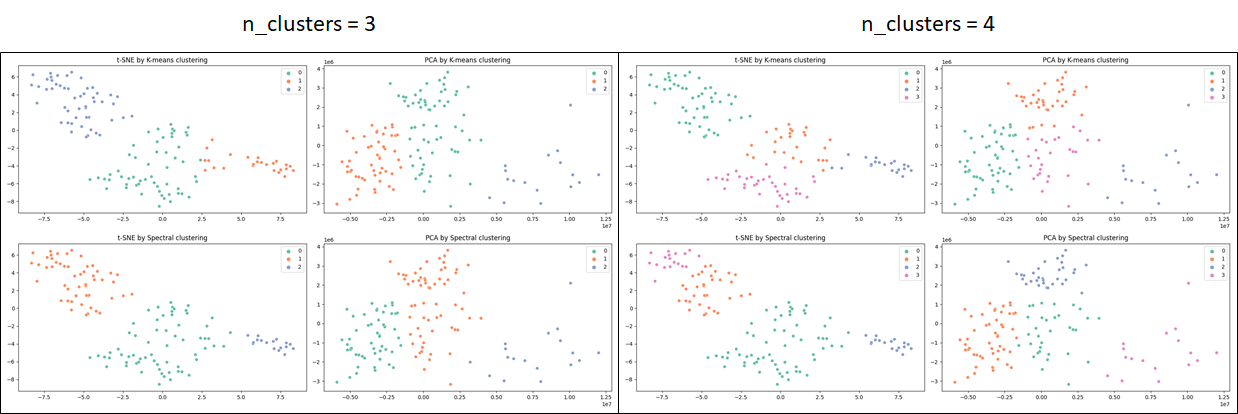
# X.rows = samples, X.columns = features
import pandas as pd
from sklearn.preprocessing import StandardScaler
from sklearn.manifold import TSNE
from sklearn.decomposition import PCA, IncrementalPCA, KernelPCA, SparsePCA
from sklearn.cluster import KMeans, SpectralClustering
import umap
import umap.plot
from umap.parametric_umap import ParametricUMAP
import matplotlib.pyplot as plt
import seaborn as sns
def plot_cluster(event, X, pheno):
n_clusters = 2
tsne = TSNE(n_components=2, random_state=0, perplexity=30).fit_transform(X) # perplexity는 이웃을 포함하는 정도
df_tsne = pd.DataFrame(tsne)
pca = PCA(n_components=2, random_state=0).fit_transform(X)
df_pca = pd.DataFrame(pca)
ump = umap.UMAP(n_components=2, random_state=0).fit_transform(X)
df_ump = pd.DataFrame(ump)
emb = ParametricUMAP(n_epochs = 500, verbose=True).fit_transform(X)
df_emb = pd.DataFrame(emb)
dnames = [df_tsne, df_pca, df_ump]#, df_emb]
cname1 = ['t-SNE','PCA','UMAP']
cname2 = [' by K-means clustering', ' by Spectral clustering']
tnames = [cname+cname2[0] for cname in cname1]+[cname+cname2[1] for cname in cname1]
fig, ax = plt.subplots(3,3,figsize=(24,14))
ax = ax.flatten()
for i, df in enumerate(dnames):
df['pheno'] = pheno
df['kmeans'] = KMeans(n_clusters=n_clusters).fit(df).labels_
df['spectral'] = SpectralClustering(n_clusters=n_clusters, affinity='nearest_neighbors', assign_labels='kmeans').fit_predict(df)
sct = sns.scatterplot(x=0, y=1, data=df, hue='pheno', palette=color, ax=ax[i])
sct.set(title=cname1[i],xlabel=None, ylabel=None)
sct.legend(title=None)
sct = sns.scatterplot(x=0, y=1, hue='kmeans', data=df, palette="Set2", ax=ax[i+3])
sct.set(title=tnames[i],xlabel=None, ylabel=None)
sct.legend(title=None)
sct = sns.scatterplot(x=0, y=1, hue='spectral', data=df, palette="Set2", ax=ax[i+6])
sct.set(title=tnames[i+3],xlabel=None, ylabel=None)
sct.legend(title=None)
plt.suptitle('[ {} ]'.format(event), y=0.985, fontweight='bold', fontsize=18)
plt.tight_layout()
plt.legend()
plt.savefig('clustering.png')
plt.clf()
df_emb['pheno'] = pheno
sct = sns.scatterplot(x=0, y=1, data=df_emb, hue='pheno', palette=color)
sct.set(title='UMAP',xlabel=None, ylabel=None)
sct.legend(title=None)
plt.suptitle(event)
plt.tight_layout()
plt.legend()
plt.savefig('UMAP.png')
plt.clf()scaler = StandardScaler()
X = scaler.fit_transform(X)
plt.style.use('seaborn-dark-palette')
plt.style.use('ggplot')
df_pca['color'] = np.where(df_pca.pheno == 'CTRL', '#008bfb', '#ff004f')
Reference
- https://en.wikipedia.org/wiki/T-distributed_stochastic_neighbor_embedding
- https://en.wikipedia.org/wiki/Principal_component_analysis
- https://en.wikipedia.org/wiki/K-means_clustering
- https://en.wikipedia.org/wiki/Spectral_clustering
- https://umap-learn.readthedocs.io/en/latest/
- https://umap-learn.readthedocs.io/en/latest/api.html#umap
'Tools' 카테고리의 다른 글
| Clustering - Fuzzy (0) | 2021.06.18 |
|---|---|
| VCF reheader (0) | 2021.06.17 |
| XGBR (0) | 2021.06.07 |
| SHAP - impact (0) | 2021.06.06 |
| Excel to tsv (0) | 2021.05.19 |
댓글