Graph vs. SMILES
- SMILES : 유사한 분자가 매우 다른 smiles로 표현됨. (학습에 어려움이 가중됨)
- Graph가 smiles보다 분자를 표현할 수 있어 보다 자연스러운 representation.

분자 그래프
- 원자 -> node, 공유결합 -> edge
- 원자와 공유결합 정보를 node와 edge에 vector로 표현.
- 모델 학습에 필요한 다양한 정보를 표현 가능.

순차적 분자그래프 생성모델
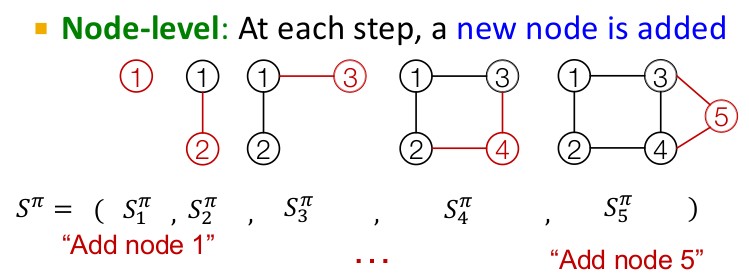
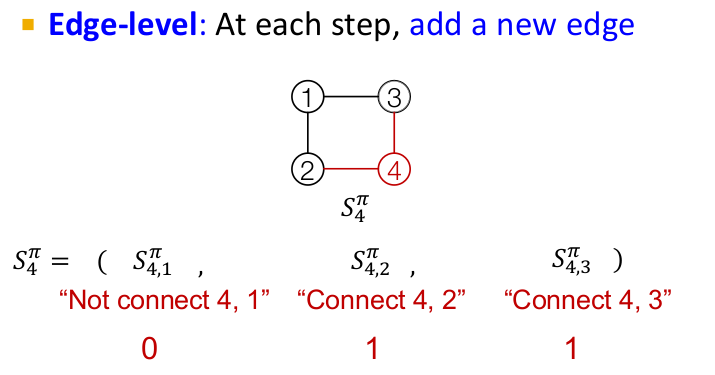
- Fixed order 또는 random order 로 분자생성. 경험적으로 차이없음.
Fragment based molecule generation using Language Model
- 사람의 관점에서 분자는 substructure의 집합.
- 원자보다는 작용기 (fragment) 단위에서 분자를 인식.
- 실용적으로 분자생성 과정이 단축되어 모델 학습이 수월해짐.
BRICS decompose

Training
- Fragment단위로 다음 fragment 선택.
- 처음과 마지막은 반드시 *가 하나여야함.

Fragment masking
필요한 이유
- 가능한 fragment가 매우 많음. (50만개 -> 수만개)
- Fragment들의 개수 분포가 매우 편중되어 있음.
방법
- 특정 frequency이하 fragment를 masking함.
- 3_1 codon이 선택되면, 이 codon에 속하는 fragment를 random하게 선택.
- 이를 통해 vocabulary size를 크게 줄일수 있음 -> 학습 속도 향상.
Fragment based molecule generation using Graph model
- LM기반 모델의 경우 고정된 vocabulary를 사용.
- Vocabulary를 neural network를 통해 embedding으로 대체.
- Vocabulary를 제한할 필요가 없음.
- Negative sampling을 통해서 fragment library size를 효율적으로 줄임.
Negative sampling
- Input fragment가 들어오면 fragment list에서 가능한 fragment를 고르는 방식은 list가 많을수록 시간이 많이 걸림.
- 그래서 list에서 random하게 k개를 선택하여 확인.
- 주어진 fragment에 새로운 fragment를 추가함. 이 과정을 반복하여 최종분자가 만들어짐.
- AI모델은 추가되어야할 fragment를 선택하고, 선택된 fragment의 어떤 부분을 연결해야할지를 결정.
- 추가해야할 fragment가 없으면 학습종료.
Junction Tree VAE
- 분자를 정해진 substructure들의 집합으로 생각.
- 1차적으로 tree 구조를 생성하고, 생성된 tree 구조로부터 분자를 생성.


Reference
- Graph기반 deep generative model을 이용한 분자 디자인1ㅣ임재창, https://youtu.be/KRvIxPHv6wc
- Graph Seminar, https://github.com/jinseock95/Graph-Seminar
- CS224W: Machine Learning with Graphs, Stanford / Fall 2019, https://snap.stanford.edu/class/cs224w-2019/
- https://github.com/snap-stanford/cs224w-notes
'Drug' 카테고리의 다른 글
| Molecule design using Deep generative models (0) | 2022.01.04 |
|---|---|
| Molecule design using Graph model 2 (0) | 2022.01.04 |
| Molecule design using SMILES (0) | 2022.01.04 |
| [Company] Standigm (0) | 2021.12.30 |
| [DRUG] Reference (0) | 2021.12.13 |




댓글