강화학습
- 시나리오 1 : 분자생성 (agent) -> 물성개성 -> reward (environment) -> 동일한 상황에서 agent는 같은 분자를 만들어낼 확률이 높아짐.
- 시나리오 2 : 분자생성 (agent) -> 물성 악화 -> penalty (environment) -> 동일한 상황에서 agent는 같은 분자를 만들어낼 확률이 낮아짐.
- Deep reinforcement learning for de novo drug design, https://doi.org/10.1126/sciadv.aap7885
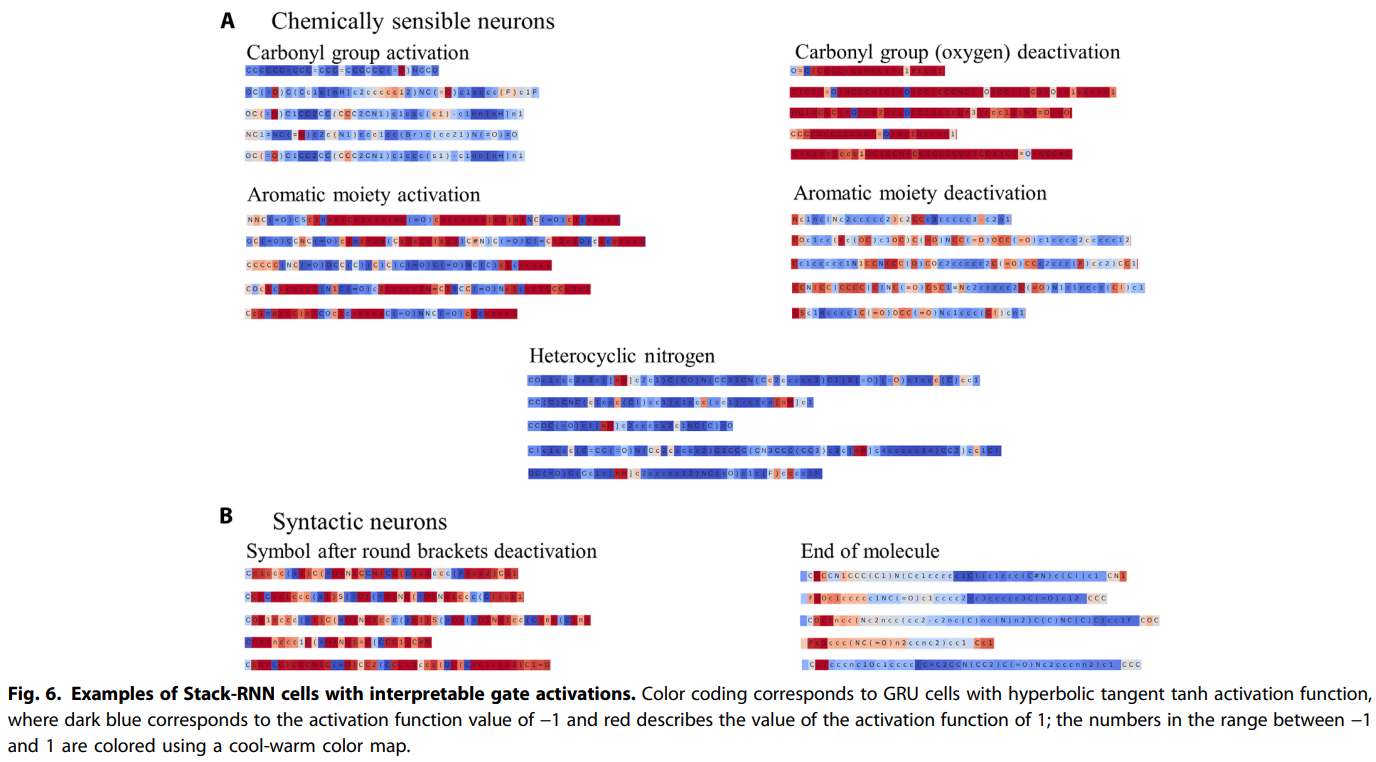
> Tanh activation 값을 통해서 해석 가능성을 보여줌. (chemically sensible groups, syntactic groups)
화학반응기반 분자 생성 모델
- 분자생성 모델을 통해 생성한 분자들이 종종 합성이 불가능.
- 분자를 사전에 정의된 reactant_reaction으로 봄.
- 이를 통해 분자를 합성 가능한 space에서 탐색하게 됨.
- Learning To Navigate The Synthetically Accessible Chemical Space Using Reinforcement Learning, https://arxiv.org/abs/2004.12485
- Reactant 개수가 매우 많음. (~수십만)
> 선택된 template이 적용 가능한 reactant만을 선별
> Agent의 action의 k-nearest reactant를 선정.
- 처음에는 invalid한 template만 선택되어 화학반응이 진행되지 않음.
> template에 masking을 해서 유효한 template만 선택되도록 함.
- 15개의 uni+82개의 bi molecule reaction template를 사용.
> Uni의 경우 reactant partner를 선택하지 않음.
GENTRL
- Generative Tensorial Reinforcement Learning (GENTRL), https://github.com/insilicomedicine/GENTRL
- Deep learning enables rapid identification of potent DDR1 kinase inhibitors, https://doi.org/10.1038/s41587-019-0224-x
- Variational autoencoder를 이용해서 latent space를 생성.
- 분자생성 및 filtering 과정.
1) 30만개 분자 생성.
2) 12,147개 분자 선별.
3) MW, LogP, TPSA등의 descriptor로 선별.
4) 150개 in-house MCF를 이용해서 filtering.
5) 남은 분자 7912개.
6) Clustering.
7) 3개 SOM으로 filtering.
8) Pharmacophore analysis.
9) Sammon mapping을 통한 분자선별.
10) 최종 40개 분자 합성.
- 실험과정
1) In vitro activity assay
2) Cell-culture activity assay
3) Cell-culture fibrosis assay
4) Cytochrome inhibition
5) Microsomal stability
6) Buffer stability
7) Phamacokinetic studies
- 결과

1) 1,2번이 좋은 potency와 selectivity를 보임.
2) 추가 실험결과 1번이 보다 적합한 것을 확인.
Limitation
- 인공지능을 통해 도출한 화합물이 학습에 사용한 약물(Ponatinib)와 구조적으로 매우 유사.
- Novel target에 대한 신규약물 발굴의 어려움.
- 이유로는 학습한 데이터가 많지 않기 때문에 유사한 구조가 생성되는 문제가 있음.
- 순수 데이터 방법만으로는 해결하기 쉽지 않은 문제.
- Biological test는 학습단계에서 고려되지 않음. 해당 과정 통과는 AI로 인한 효과가 아님.
Reference
- The applications of deep generative modelsㅣ임재창, https://youtu.be/vGojaIKA7V4
'Drug' 카테고리의 다른 글
| ZINC database (0) | 2022.01.10 |
|---|---|
| Molecule design using Graph model 2 (0) | 2022.01.04 |
| Molecule design using Graph model 1 (0) | 2022.01.04 |
| Molecule design using SMILES (0) | 2022.01.04 |
| [Company] Standigm (0) | 2021.12.30 |



댓글